DNA sequencing
序
开学一周,一直提上日程的测序简史,也没有时间去好好的落实。于是乎,我鼓鼓勇气,尝试着去把这段从1977年到2020年的漫长而又渺小的四十几年说的有趣些儿。
当我起笔去写这篇文章的时候,我还在晒太阳,关于测序简史这一块,我一直不知道怎么去娓娓道来,不知道如何才能说得清楚,硬着头皮利用自己知道的皮毛和搜刮来的知识给大家编织一个我所认为的测序简史。
由于这篇文章内容过长,第一次尝试用MD语法写文章,熟悉一下,言归正传。
啥叫测序?这个官方有官方的解释,大家可以自行百度,我觉得通俗意义上跟测序身高体重三围没啥区别,区别在于测序难度大并且包含的信息量大。
身高仪测量你的身高信息,体重称量你的体重信息,不是健康信息。测序是测量你的遗传信息。
遗传信息,大家应该都清楚,如果不清楚的话麻烦各位翻一翻高中的肺炎双球菌实验,讲的就是啥是遗传信息,如何发现遗传信息的。
放张图,方便大家回忆。

原来的科学家们通过老鼠死没死,最终得到的结论是DNA是主要的遗传物质,部分物种的遗传物质是RNA。
在弄清楚这个事情之后,大家也都知道沃森和克里克还有一些被遗忘的科学家一起努力弄清楚了DNA是双螺旋结构。并且(A-T,G-C)。
第一代测序技术
1、简介
第一代DNA测序技术用的是1975年由桑格(Sanger)和考尔森(Coulson)开创的链终止法或者是1976-1977年由马克西姆(Maxam)和吉尔伯特(Gilbert)发明的化学法(链降解). 并在1977年,桑格测定了第一个基因组序列,是噬菌体X174的,全长5375个碱基。自此,人类获得了窥探生命遗传差异本质的能力,并以此为开端步入基因组学时代。研究人员在Sanger法的多年实践之中不断对其进行改进,在2001年,完成的首个人类基因组图谱就是以改进了的Sanger法为其测序基础。
一代测序技术的原理见下图。在模板中首先分别加入A、T、G、C和四种ddNTP双脱氧核苷酸(加入ddNTP序列合成会终止),如下图第一个加入ddATP,这样每一个位置上的A位置会大量的被ddATP替代,然后终止,然后再分别加入其他的ddNTP,让他随机终止。这样对得到的这些序列进行跑胶。就得到了如下的胶图。根据ACGT的加入顺序和位置,获取信息。这个方法准确率高,费用高,是先合成,再测序的。

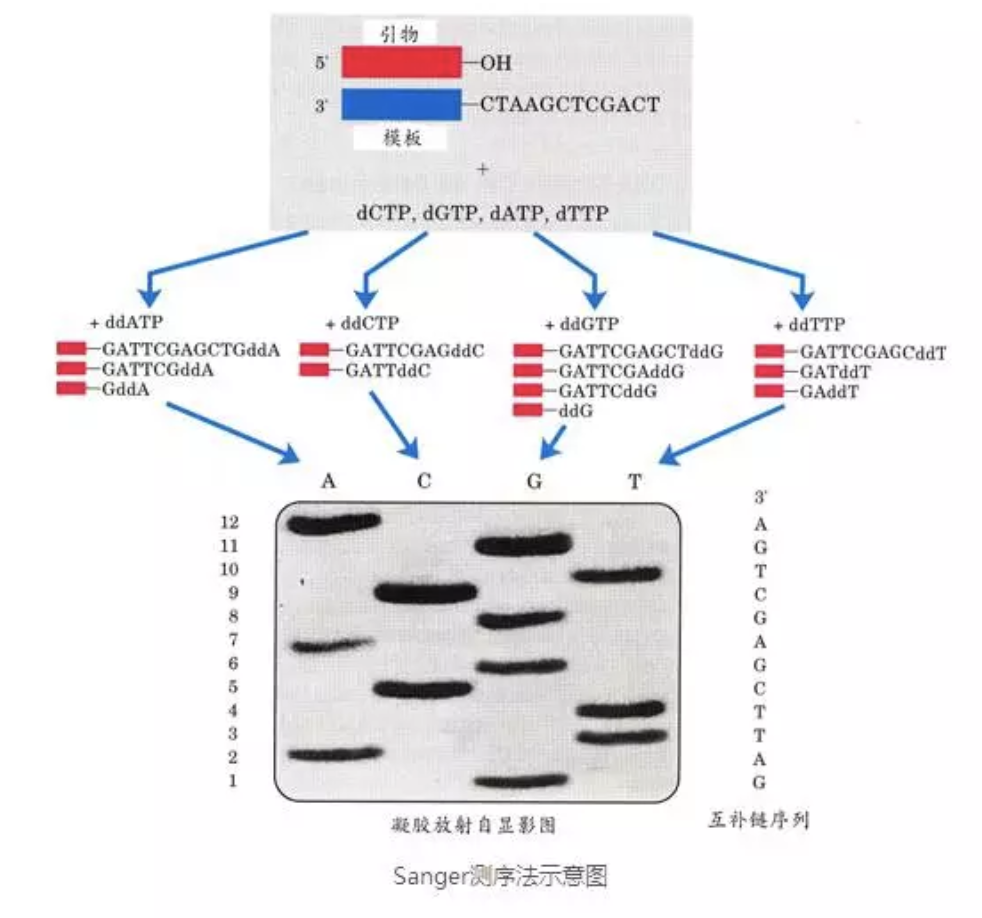 桑格先生13年与世长辞,但是一代测序技术在他发明之后经过各个单位的改进,今天还被大量使用。
桑格先生13年与世长辞,但是一代测序技术在他发明之后经过各个单位的改进,今天还被大量使用。
NCBI的悼文:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3903207/
Of the three main activities involved in scientific research, thinking, talking, and doing, I much prefer the last and am probably best at it. I am all right at the thinking, but not much good at the talking.
—Frederick Sanger, 1988
2、一代测序主要应用方向
高通量测序最近这几年很火越来越火,但是世界上更多的还是一帮天天做分子克隆、养细胞、养细菌、杂蛋白的生物学家,究其原因Sanger测序还是测序届的金标准,由于精确度高于2、3代测序且保持大白菜价格使之地位稳固。
应用范围:De Novo测序、重测序: 如突变检测、SNPs、插入、缺失克隆产物验证、比较基因组、分型: 如微生物和真菌鉴定、HLA分型、病毒分型。
其它: 如甲基化分析(重亚硫酸盐测序)和SAGE(基因表达串联分析)方法
临床应用:肿瘤突变基因的检测和肿瘤个体化治疗。
第⼆代测序技术
⼆代测序技术主要有三家公司,罗⽒的454技术,illumina的Hiseq和 Solexa技术还有ABI的Solid技术。不管是哪家公司,其具体原理如何,暂且不说。他们都 是边合成边测序,也就是说通过在序列合成的同时通过各种标记进⾏实时的序列识别。
由于⼆代测序需要对荧光信号进⾏识别,但是由于荧光信号较弱,因此需要进⾏扩增建 库。也就是这⼀步导致⼆代测序存在偏好性。
应用方向:⼆代测序⽬前是科研市场上的主⼒,⼴泛的使⽤在物种基因组测序,转录组测序,群体测序。另外这两年也在寻求医学上的发展,随着成本的降低,其在医学市场上的应⽤将会越来越多。
1、二代测序相关名词解释
什么是⾼通量测序?
⾼通量测序技术(High-throughputsequencing,HTS)是对传统Sanger测序(称为⼀代测序技术)⾰命性的改变, ⼀次对⼏⼗万到⼏百万条核酸分⼦进⾏序列测定, 因此在有些⽂献 中称其为下⼀代测序技术(next generation sequencing,NGS )⾜见其划时代的改变, 同时 ⾼通量测序使得对⼀个物种的转录组和基因组进⾏细致全貌的分析成为可能, 所以又被称为深度测序(Deep sequencing)。
什么是基因组重测序(Genome Re-sequencing)
全基因组重测序是对基因组序列已知的个体进⾏基因组测序,并在个体或群体⽔平上进⾏ 差异性分析的⽅法。随着基因组测序成本的不断降低,⼈类疾病的致病突变研究由外显⼦ 区域扩⼤到全基因组范围。通过构建不同长度的插⼊⽚段⽂库和短序列、双末端测序相结 合的策略进⾏⾼通量测序,实现在全基因组⽔平上检测疾病关联的常见、低频、甚⾄是罕见的突变位点,以及结构变异等,具有重⼤的科研和产业价值。
什么是de novo测序
de novo测序也称为从头测序:其不需要任何现有的序列资料就可以对某个物种进⾏测序, 利⽤⽣物信息学分析⼿段对序列进⾏拼接,组装,从⽽获得该物种的基因组图谱。获得⼀ 个物种的全基因组序列是加快对此物种了解的重要捷径。随着新⼀代测序技术的飞速发 展,基因组测序所需的成本和时间较传统技术都⼤⼤降低,⼤规模基因组测序渐⼊佳境, 基因组学研究也迎来新的发展契机和⾰命性突破。利⽤新⼀代⾼通量、⾼效率测序技术以 及强⼤的⽣物信息分析能⼒,可以⾼效、低成本地测定并分析所有⽣物的基因组序列。
什么是外显⼦测序(whole exon sequencing)
外显⼦组测序是指利⽤序列捕获技术将全基因组外显⼦区域DNA捕捉并富集后进⾏⾼通量 测序的基因组分析⽅法。外显⼦测序相对于基因组重测序成本较低,对研究已知基因的 SNP、Indel等具有较⼤的优势,但⽆法研究基因组结构变异如染⾊体断裂重组等。
什么是mRNA测序 (RNA-seq)
转录组学(transcriptomics)是在基因组学后新兴的⼀门学科,即研究特定细胞在某⼀功 能状态下所能转录出来的所有RNA(包括mRNA和⾮编码RNA)的类型与拷贝数。 Illumina提供的mRNA测序技术可在整个mRNA领域进⾏各种相关研究和新的发现。 mRNA测序不对引物或探针进⾏设计,可⾃由提供关于转录的客观和权威信息。研究⼈员仅需要⼀次试验即可快速⽣成完整的poly-A尾的RNA完整序列信息,并分析基因表达、 cSNP、全新的转录、全新异构体、剪接位点、等位基因特异性表达和罕见转录等最全⾯的 转录组信息。简单的样品制备和数据分析软件⽀持在所有物种中的mRNA测序研究。
什么是small RNA测序
SmallRNA(micro RNAs、siRNAs和 pi RNAs)是⽣命活动重要的调控因⼦,在基因表达调控、⽣物个体发育、代谢及疾病的发⽣等⽣理过程中起着重要的作⽤。Illumina能够对 细胞或者组织中的全部Small RNA进⾏深度测序及定量分析等研究。实验时⾸先将18-30 nt 范围的Small RNA从总RNA中分离出来,两端分别加上特定接头后体外反转录做成cDNA 再做进⼀步处理后,利⽤测序仪对DNA⽚段进⾏单向末端直接测序。通过Illumina对SmallRNA⼤规模测序分析,可以从中获得物种全基因组⽔平的miRNA图谱,实现包括新 miRNA分⼦的挖掘,其作⽤靶基因的预测和鉴定、样品间差异表达分析、miRNAs聚类和 表达谱分析等科学应⽤。
什么是miRNA测序
成熟的microRNA(miRNA)是17~24nt的单链⾮编码RNA分⼦,通过与mRNA相互作⽤ 影响⽬标mRNA的稳定性及翻译,最终诱导基因沉默,调控着基因表达、细胞⽣长、发育 等⽣物学过程。基于第⼆代测序技术的microRNA测序,可以⼀次性获得数百万条 microRNA序列,能够快速鉴定出不同组织、不同发育阶段、不同疾病状态下已知和未知 的microRNA及其表达差异,为研究microRNA对细胞进程的作⽤及其⽣物学影响提供了有 ⼒⼯具。
什么是Chip-seq
染⾊质免疫共沉淀技术(ChromatinImmunoprecipitation,ChIP)也称结合位点分析法, 是研究体内蛋⽩质与DNA相互作⽤的有⼒⼯具,通常⽤于转录因⼦结合位点或组蛋⽩特异 性修饰位点的研究。将ChIP与第⼆代测序技术相结合的ChIP-Seq技术,能够⾼效地在全基 因组范围内检测与组蛋⽩、转录因⼦等互作的DNA区段。
ChIP-Seq的原理是:⾸先通过染⾊质免疫共沉淀技术(ChIP)特异性地富集⽬的蛋⽩结合 的DNA⽚段,并对其进⾏纯化与⽂库构建;然后对富集得到的DNA⽚段进⾏⾼通量测序。 研究⼈员通过将获得的数百万条序列标签精确定位到基因组上,从⽽获得全基因组范围内 与组蛋⽩、转录因⼦等互作的DNA区段信息。
什么是CHIRP-Seq
CHIRP-Seq( Chromatin Isolationby RNA Purification )是⼀种检测与RNA绑定的DNA和蛋 ⽩的⾼通量测序⽅法。⽅法是通过设计⽣物素或链霉亲和素探针,把⽬标RNA拉下来以 后,与其共同作⽤的DNA染⾊体⽚段就会附在到磁珠上,最后把染⾊体⽚段做⾼通量测 序,这样会得到该RNA能够结合到在基因组的哪些区域,但由于蛋⽩测序技术不够成熟, ⽆法知道与该RNA结合的蛋⽩。
什么是RIP-seq
RNA Immunoprecipitation是研究细胞内RNA与蛋⽩结合情况的技术,是了解转录后调控 ⽹络动态过程的有⼒⼯具,能帮助我们发现miRNA的调节靶点。这种技术运⽤针对⽬标蛋 ⽩的抗体把相应的RNA-蛋⽩复合物沉淀下来,然后经过分离纯化就可以对结合在复合物上 的RNA进⾏测序分析。
RIP可以看成是普遍使⽤的染⾊质免疫沉淀ChIP技术的类似应⽤,但由于研究对象是RNA蛋⽩复合物⽽不是DNA-蛋⽩复合物,RIP实验的优化条件与ChIP实验不太相同(如复合物 不需要固定,RIP反应体系中的试剂和抗体绝对不能含有RNA酶,抗体需经RIP实验验证等 等)。RIP技术下游结合microarray技术被称为RIP-Chip,帮助我们更⾼通量地了解癌症 以及其它疾病整体⽔平的RNA变化。
什么是CLIP-seq
CLIP-seq,又称为HITS-CLIP,即紫外交联免疫沉淀结合⾼通量测序(crosslinkingimmunprecipitationand high-throughput sequencing), 是⼀项在全基因组⽔平揭⽰RNA分 ⼦与RNA结合蛋⽩相互作⽤的⾰命性技术。其主要原理是基于RNA分⼦与RNA结合蛋⽩在 紫外照射下发⽣耦联,以RNA结合蛋⽩的特异性抗体将RNA-蛋⽩质复合体沉淀之后,回 收其中的RNA⽚段,经添加接头、RT-PCR等步骤,对这些分⼦进⾏⾼通量测序,再经⽣ 物信息学的分析和处理、总结,挖掘出其特定规律,从⽽深⼊揭⽰RNA结合蛋⽩与RNA分 ⼦的调控作⽤及其对⽣命的意义。
什么是染⾊体构象捕获技术
3C 通常是⽤启动⼦或者某⼀个基因或者基因组某⼀个短的⽚段在邻近的⼏⼗kb或者⼏百kb 基因组扫描可以获得相互作⽤区域。由于实验需要特异性引物,因⽽实验室相当费⼒的, 且检测范围⼩。
4C同3C⼀样做单位点的检测,但其检测扩展到了整个基因组上。主要是引⼊了反向PCR, 因⽽只需要对这⼀单⼀位点设计引物即可。
5C 做两个⼤⽚段之间相互作⽤点的检测,可以达到10Mb⽔平。其仍需使⽤引物,且引物设计是其技术的难点。
Hi-C 可以实现基因组对基因组⽔平的检测,但是获得⾼精度需要⾮常⼤的测序深度
ChIA-PET标在于特定的蛋⽩因⼦及其相关联的染⾊质相互作⽤.该技术将配对末端标签测序技术与ChIP相结合, 对富集了某种蛋⽩质的DNA ⽚段进⾏交联, 可以测定全基因组范围的特定转录因⼦参与的染⾊质远程交互作⽤, 从⽽可以呈现⾼特异性和⾼分辨率的染⾊质相 互作⽤.什么是Hi-C辅助基因组组装
Hi-C辅助基因组组装是指在已有⼆代或三代或光学图谱辅助组装的Draft genome序列和已 知染⾊体数⽬的前提下,利⽤Hi-C测序数据将Draft genome序列进⾏染⾊体群组的划分, 并确定各序列在染⾊体上的顺序和⽅向,使基因组组装组装⽔平提升到染⾊体⽔平的技 术。
什么是metagenomic(宏基因组)
Magenomics研究的对象是整个微⽣物群落。相对于传统单个细菌研究来说,它具有众多优 势,其中很重要的两点:
(1)微⽣物通常是以群落⽅式共⽣于某⼀⼩⽣境中,它们的很多特性是基于整个群落环境及个体间的相互影响的,因此做Metagenomics研究⽐做单个个体的研究更能发现其特性;
(2)Metagenomics研究⽆需分离单个细菌,可以研究那些不能被实验室分离培养的微⽣物。
宏基因组是基因组学⼀个新兴的科学研究⽅向。宏基因组学(又称元基因组学,环境基因组学,⽣态基因组学等),是研究直接从环境样本中提取的基因组遗传物质的学科。传统的微⽣物研究依赖于实验室培养,宏基因组的兴起填补了⽆法在传统实验室中培养的微⽣物研究的空⽩。过去⼏年中,DNA测序技术的进步以及测序通量和分析⽅法的改进使得⼈们得以⼀窥这⼀未知的基因组科学领域。
什么是SNP、SNV(单核苷酸位点变异)
单核苷酸多态性singlenucleotide polymorphism,SNP 或单核苷酸位点变异SNV。个体间 基因组DNA序列同⼀位置单个核苷酸变异(替代、插⼊或缺失)所引起的多态性。不同物 种、个体基因组DNA序列同⼀位置上的单个核苷酸存在差别的现象。有这种差别的基因 座、DNA序列等可作为基因组作图的标志。⼈基因组上平均约每1000个核苷酸即可能出现 1个单核苷酸多态性的变化,其中有些单核苷酸多态性可能与疾病有关,但可能⼤多数与疾 病⽆关。单核苷酸多态性是研究⼈类家族和动植物品系遗传变异的重要依据。在研究癌症 基因组变异时,相对于正常组织,癌症中特异的单核苷酸变异是⼀种体细胞突变(somatic mutation),称做SNV。
什么是INDEL (基因组⼩⽚段插⼊)
基因组上⼩⽚段(>50bp)的插⼊或缺失,形同SNP/SNV。
什么是copy number variation(CNV):基因组拷贝数变异
基因组拷贝数变异是基因组变异的⼀种形式,通常使基因组中⼤⽚段的DNA形成⾮正常的 拷贝数量。例如⼈类正常染⾊体拷贝数是2,有些染⾊体区域拷贝数变成1或3,这样,该区 域发⽣拷贝数缺失或增加,位于该区域内的基因表达量也会受到影响。如果把⼀条染⾊体 分成A-B-C-D四个区域,则A-B-C-C-D/A-C-B-C-D/A-C-C-B-C-D/A-B-D分别发⽣了C区域 的扩增及缺失,扩增的位置可以是连续扩增如A-B-C-C-D也可以是在其他位置的扩增,如 A-C-B-C-D。
什么是structure variation(SV):基因组结构变异
染⾊体结构变异是指在染⾊体上发⽣了⼤⽚段的变异。主要包括染⾊体⼤⽚段的插⼊和缺 失(引起CNV的变化),染⾊体内部的某块区域发⽣翻转颠换,两条染⾊体之间发⽣重组 (inter-chromosometrans-location)等。⼀般SV的展⽰利⽤Circos软件。
什么是Segment duplication
⼀般称为SD区域,串联重复是由序列相近的⼀些DNA⽚段串联组成。串联重复在⼈类基因 多样性的灵长类基因中发挥重要作⽤。在⼈类染⾊体Y和22号染⾊体上,有很⼤的SD序 列。
什么是genotype and phenotype
既基因型与表型;⼀般指某些单核苷酸位点变异与表现形式间的关系。
什么是Read?
⾼通量测序平台产⽣的短序列就称为reads。PE125,就是读长为125bp双端测序。
什么是Contig?
拼接软件基于reads之间的overlap区,拼接获得的序列称为Contig(重叠群),⽆N。
什么是Scaffold?
基因组de novo测序,通过reads拼接获得Contigs后,往往还需要构建454 Paired-end库或 Illumina Mate-pair库,以获得⼀定⼤⼩⽚段(如3Kb、6Kb、10Kb、20Kb)两端的序 列。基于这些序列,可以确定⼀些Contig之间的顺序关系,这些先后顺序已知的Contigs组 成Scaffold(含有N)
什么是Contig N50?
Reads拼接后会获得⼀些不同长度的Contigs。将所有的Contig长度相加,能获得⼀个 Contig总长度。然后将所有的Contigs按照从长到短进⾏排序,如获得Contig 1,Contig 2, Contig 3...………Contig 25。将Contig按照这个顺序依次相加,当相加的长度达到Contig总 长度的⼀半时,最后⼀个加上的Contig长度即为Contig N50。举例:Contig 1+Contig 2+ Contig 3+Contig4=Contig总长度*1/2时,Contig 4的长度即为Contig N50。Contig N50可以作为基因组拼接的结果好坏的⼀个判断标准。
什么是Scaffold N50?
Scaffold N50与Contig N50的定义类似。Contigs拼接组装获得⼀些不同长度的Scaffolds。 将所有的Scaffold长度相加,能获得⼀个Scaffold总长度。然后将所有的Scaffolds按照从长 到短进⾏排序,如获得Scaffold 1,Scaffold 2,Scaffold 3...………Scaffold 25。将Scaffold 按照这个顺序依次相加,当相加的长度达到Scaffold总长度的⼀半时,最后⼀个加上的 Scaffold长度即为Scaffold N50。举例:Scaffold 1+Scaffold 2+Scaffold 3 +Scaffold 4 +Scaffold 5=Scaffold总长度*1/2时,Scaffold 5的长度即为Scaffold N50。Scaffold N50可 以作为基因组拼接的结果好坏的⼀个判断标准。
什么是测序深度和覆盖度?
测序深度是指测序得到的总碱基数与待测基因组⼤⼩的⽐值。假设⼀个基因⼤⼩为2M,测 序深度为10X,那么获得的总数据量为20M。覆盖度是指测序获得的序列占整个基因组的⽐例。由于基因组中的⾼GC、重复序列等复杂结构的存在,测序最终拼接组装获得的序列 往往⽆法覆盖有所的区域,这部分没有获得的区域就称为Gap。例如⼀个细菌基因组测 序,覆盖度是98%,那么还有2%的序列区域是没有通过测序获得的。
什么是RPKM、FPKM
RPKM,ReadsPer Kilobase of exon model per Million mapped reads, is defined in thisway [Mortazavi etal., 2008]:
每1百万个map上的reads中map到外显⼦的每1K个碱基上的reads个数。
假如有1百万个reads映射到了⼈的基因组上,那么具体到每个外显⼦呢,有多少映射上了 呢,⽽外显⼦的长度不⼀,那么每1K个碱基上又有多少reads映射上了呢,这⼤概就是这个 RPKM的直观解释。
如果对应特定基因的话,那么就是每1000000 mapped到该基因上的reads中每kb有多少是 mapped到该基因上的exon的read
Total exon reads
This is the number in the column with header Total exonreads in the rowfor the gene. This is the number of reads that have beenmapped to a region inwhich an exon is annotated for the gene or across theboundaries of two exons oran intron and an exon for an annotated transcript ofthe gene. For eukaryotes,exons and their internal relationships are defined byannotations of type mRNA.
映射到外显⼦上总的reads个数。这个是映射到 某个区域上的reads个数,这个区域或者是已知注释的基因或者跨两个外显⼦的边界或者是 某个基因已经注释的转录本的内含⼦、外显⼦。对于真核⽣物来说,外显⼦和它们⾃⼰内 部的关系由某类型的mRNA来注释。
Exonlength:
This is the number in the column with theheader Exon length inthe row for the gene, divided by 1000. This is calculatedas the sum of thelengths of all exons annotated for the gene. Each exon isincluded only once inthis sum, even if it is present in more annotatedtranscripts for the gene.Partly overlapping exons will count with their fulllength, even though theyshare the same region.
外显⼦的长度。计算时,计算所有某个 基因已注释的所有外显⼦长度的总和。即使某个基因以多种注释的转录本呈现,这个外显 ⼦在求和时只被包含⼀次。即使部分重叠的外显⼦共享相同的区域,重叠的外显⼦以其总 长来计算。
Mapped reads
The sum of all the numbers in the column with header Totalgenereads. The Total gene reads for a gene is the total number ofreads that aftermapping have been mapped to the region of the gene. Thus thisincludes all thereads uniquely mapped to the region of the gene as well asthose of the readswhich match in more places (below the limit set in thedialog in figure 18.110) that have been allocated tothis gene's region. Agene's region is that comprised of the flanking regions(if it was specified infigure 18.110), the exons, the introns andacross exon-exonboundaries of all transcripts annotated for the gene. Thus,the sum of the totalgene reads numbers is the number of mapped reads for thesample (you can findthe number in the RNA-Seq report).
map的reads总和。映射到某个基因上的所有reads总数。因此这包含所有的唯⼀映射到这个区域上的reads。
举例:⽐如对应到该基因的read有1000个,总reads个数有100万,⽽该基因的外显⼦总长 为5kb,那么它的RPKM为:10^91000(reads个数)/10^6(总reads个数)5000(外显⼦长 度)=200或者:1000(reads个数)/1(百万)*5(K)=200这个值反映基因的表达⽔平。
FPKM(fragmentsper kilobase of exon per million fragments mapped)
FPKM与RPKM计算⽅法基本⼀致。不同点就是FPKM计算的是fragments,⽽RPKM计算 的是reads。Fragment⽐read的含义更⼴,因此FPKM包含的意义也更⼴,可以是pair-end 的⼀个fragment,也可以是⼀个read。
什么是转录本重构
⽤测序的数据组装成转录本。有两种组装⽅式:1,de-novo构建; 2,有参考基因组重 构。其中de-novo组装是指在不依赖参考基因组的情况下,将有overlap的reads连接成⼀个 更长的序列,经过不断的延伸,拼成⼀个个的contig及scaffold。常⽤⼯具包括velvet, trans-ABYSS,Trinity等。有参考基因组重构,是指先将read贴回到基因组上,然后在基因组通过reads覆盖度,junction位点的信息等得到转录本,常⽤⼯具包括scripture、 cufflinks。
什么是表达谱
基因表达谱(geneexpression profile):指通过构建处于某⼀特定状态下的细胞或组织的⾮偏性cDNA⽂库,⼤规模cDNA测序,收集cDNA序列⽚段、定性、定量分析其mRNA群体组 成,从⽽描绘该特定细胞或组织在特定状态下的基因表达种类和丰度信息,这样编制成的数据 表就称为基因表达谱
什么是⽐较基因组学
⽐较基因组学(ComparativeGenomics)是基于基因组图谱和测序基础上,对已知的基因和基因组结构进⾏⽐较,来了解基因的功能、表达机理和物种进化的学科。利⽤模式⽣物基 因组与⼈类基因组之间编码顺序上和结构上的同源性,克隆⼈类疾病基因,揭⽰基因功能 和疾病分⼦机制,阐明物种进化关系,及基因组的内在结构。
什么是基因组注释
基因组注释(Genomeannotation) 是利⽤⽣物信息学⽅法和⼯具,对基因组所有基因的⽣物学功能进⾏⾼通量注释,是当前功能基因组学研究的⼀个热点。基因组注释的研究内容包括 基因识别和基因功能注释两个⽅⾯。基因识别的核⼼是确定全基因组序列中所有基因的确切位置。
第三代测序技术
1、简介
第三代测序技术⽬前在市场上较为活跃的是Pacbio公司的RS II系列 和Sequl系列。⼆代测序主要的长度较短的问题,Pacbio将会彻底给予解决,⽬前其读长在9Kb以上,准确性在85%以上。通量较⼆代差距很⼤,⽬前Sequl的⼀个cell可以产出5 Gb左右。这个通量⽬前对于科研市场是消费的起的,但是对于临床检测,成本较⾼。
第三代测序技术是指单分⼦测序技术。DNA测序时,不需要经过PCR扩增,实现了对每⼀ 条DNA分⼦的单独测序。第三代测序技术也叫从头测序技术,即单分⼦实时DNA测序。
主要包括单分⼦荧光技术,也就是不需要扩增,每⼀个分⼦显⽰⼀种光,然后实时去监控,去读取。因此这⾥如何构建⼀个环境,让核酸分⼦单独发光,去识别是技术难点。
最近国内较⽕的瀚海基因的GenoCare也是基于单分⼦荧光技术的。
⽬前三代数据主要应⽤在科研市场的两个⽅向上,第⼀个是基因组的组装,另⼀个就是全 长转录组。
另外⼀种技术就是单分⼦纳⽶技术,顾名思义,就是让核酸分⼦单独的经过纳⽶通道,通 过每个分⼦不同的电信号进⾏识别。这个技术的代表是⽜津⼤学的naropore技术。
纳⽶孔测序技术
纳⽶孔测序技术是最近⼏年兴起的新⼀代测序技术。⽬前测序长度可以达到150kb。这项 技术开始于90年代,经历了三个主要的技术⾰新:
⼀、单分⼦DNA从纳⽶孔通过;
⼆、纳⽶孔上的酶对于测序分⼦在单核苷酸精度的控制;
三、单核苷酸的测序精度控制。⽬前市场上⼴泛接受的纳⽶孔测序平台是Oxford Nanopore Technologies(ONT)公司的MinION 纳⽶孔测仪。它的特点是单分⼦测序,测序读长长(超过150kb),测序速度快,测序数 据实时监控,机器⽅便携带等。这篇综述重点总结了MinION测序仪的技术特点和应⽤领域。
MinION测序技术简介
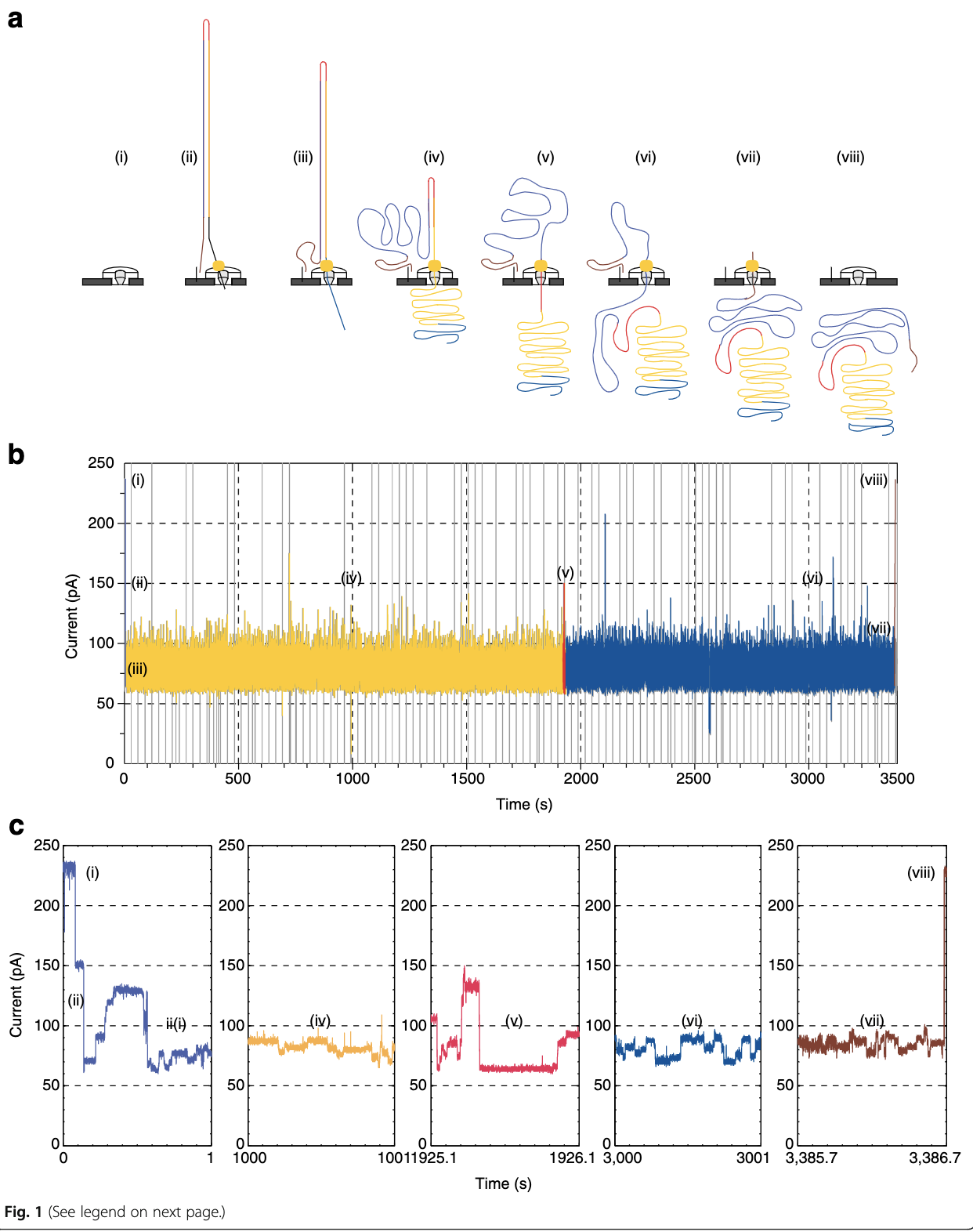
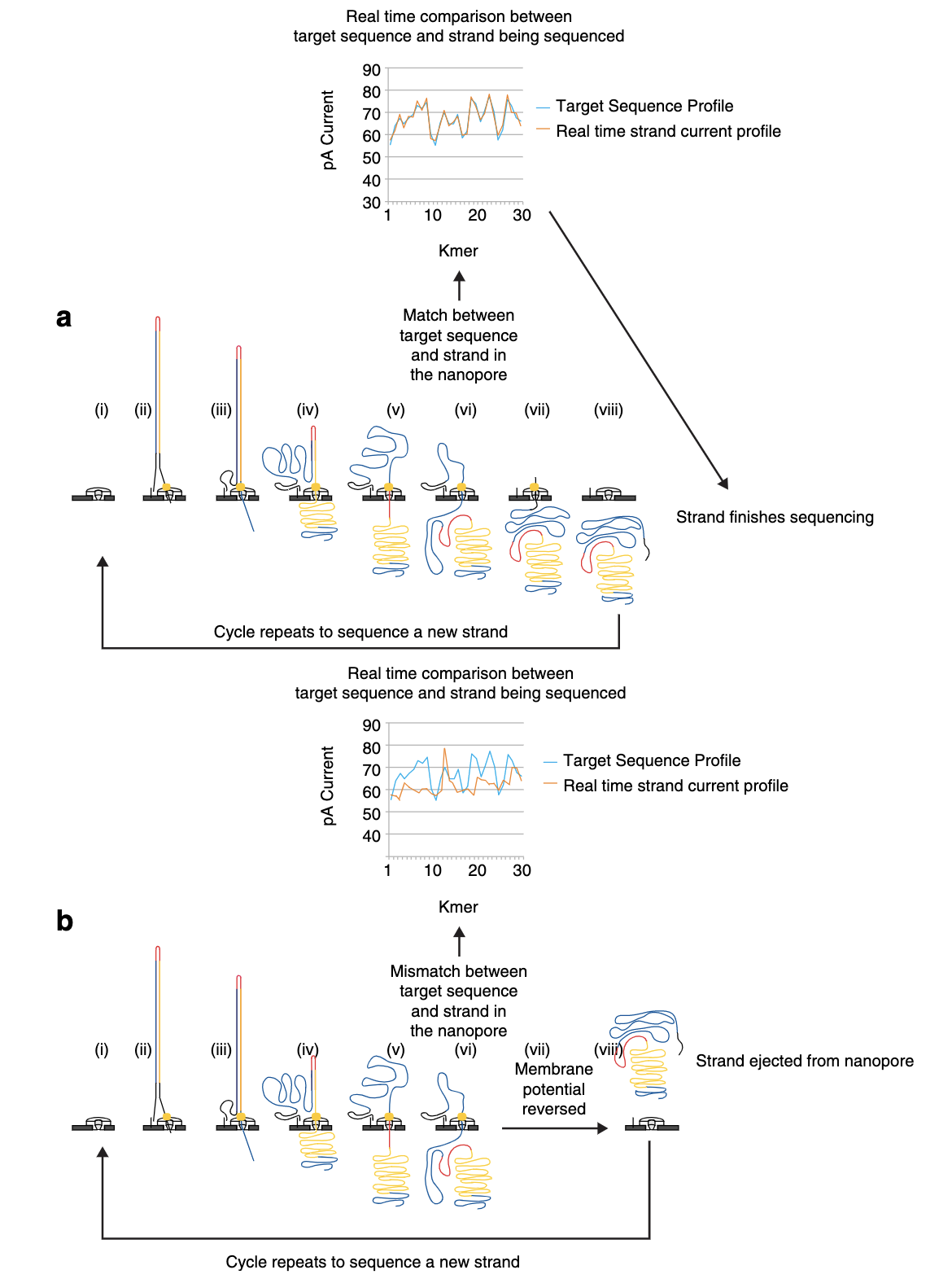
MinION纳⽶孔测序仪的核⼼是⼀个有2,048个纳⽶孔,分成512组,由专⽤集成电路控制 的flow cell。测序原理见图1a所⽰:⾸先,将双分⼦DNA连接lead adaptor(蓝⾊), hairpin adaptor(红⾊)和trailing adaptor(棕⾊);当测序开始,lead adaptor带领测序 分⼦进⼊由酶控制的纳⽶孔,lead adaptor后是template read(即待测序的DNA分⼦)通 过纳⽶孔,hairpin adaptor的作⽤是DNA双链测序的保证,然后complement read(待测序分⼦的互补链)通过纳⽶孔,最后是trailing adaptor通过。在上述测序⽅法中, template read和complement read依次通过纳⽶孔,利⽤pairwise alignment,它们组合成 2D read;⽽在另外⼀种测序⽅法中,不使⽤hairpin adaptor,只测序template read,最终 形成1D read。后⼀种测序⽅法通量更⾼,但是测序准确性低于2D read。每个接头序列(adaptor)通过纳⽶孔引起的电流变化不同(图1c),这种差别可以⽤来做碱基识别。
参考文献
The Oxford Nanopore MinION: delivery of nanopore sequencing to the genomics community
https://link.springer.com/content/pdf/10.1186/s13059-016-1103-0.pdf